Bu proje, sentetik histopatoloji görüntüleri üretmek için çeşitli derin öğrenme tekniklerini kullanır. Çalışmada, Autoencoders, GAN'ler ve difüzyon modelleri gibi yaklaşımlar denenmiştir. Ayrıca, Google'ın yayınladığı Path Foundation modeli ile görüntü embedleri elde edilmiştir.
- Veri Hazırlama
- Model Mimarileri
- Eğitim Süreci
- Sonuçlar ve Değerlendirme
- Kurulum ve Kullanım
- Referanslar
Bu çalışmada kullanılan histopatoloji görüntüleri çeşitli veri kaynaklarından elde edilmiştir. Veri ön işleme aşamaları şunlardır:
-
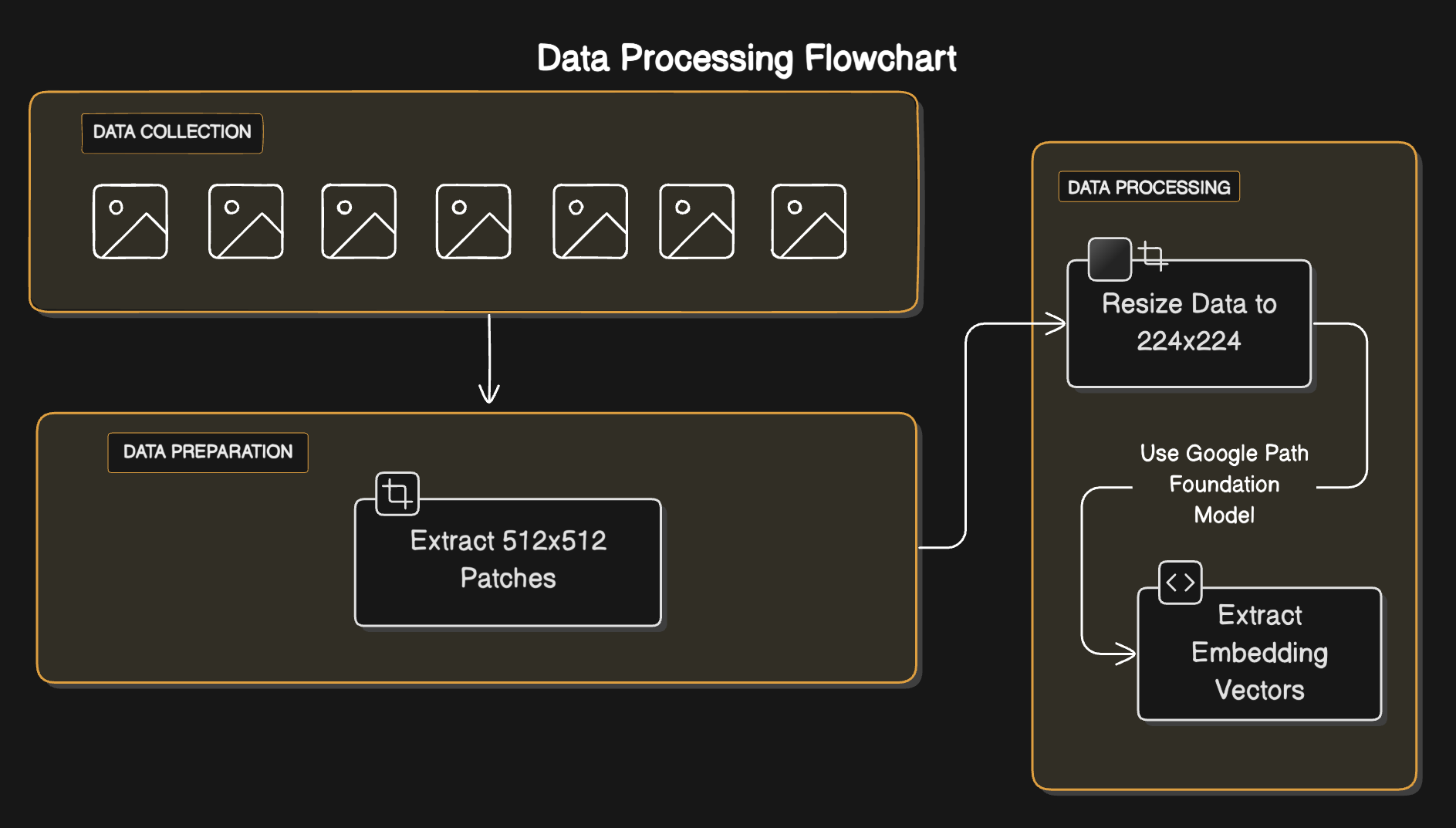
Görüntü Toplama: Histopatoloji görüntülerinin toplanması ve organizasyonu.
Veri Seti: Bu aşama CAMELYON16 , CANCER IMAGING ARCHIVE-KIDNEY, CANCER IMAGING ARCHIVE-COLON, CANCER IMAGING ARCHIVE-LUNG veri setlerinden alınan bir grup histopatoloji görüntüsünün tüm slayt görüntülerinin toplanmasını içerir. Bu kaynaklardan toplanan tüm slayt görüntülerinin listesi, aşağıdaki tabloda verilmiştir.
CAMELYON16 CANCER IMAGING ARCHIVE-KIDNEY CANCER IMAGING ARCHIVE-COLON CANCER IMAGING ARCHIVE-LUNG test_001.tifC3L-00004-21.svsMSB-00241-01-05.svsC3L-00001-21.svstest_002.tifC3L-00004-26.svsMSB-00241-01-06.svsC3L-00001-26.svstest_003.tifC3L-00010-21.svsMSB-00352-03-05.svsC3L-00009-21.svstest_004.tifC3L-00010-26.svsMSB-00352-03-10.svsC3L-00009-26.svstest_005.tifC3L-00011-21.svsMSB-00352-05-02.svsC3L-00080-21.svstest_006.tifC3L-00011-26.svsMSB-00643-03-06.svsC3L-00080-26.svstest_007.tifC3L-00026-21.svsMSB-00643-03-11.svsC3L-00083-21.svstest_008.tifC3L-00026-26.svsMSB-00643-03-12.svsC3L-00083-26.svstest_009.tifC3L-00079-21.svsMSB-00643-03-13.svsC3L-00093-21.svstest_010.tifC3L-00079-26.svsMSB-00952-01-02.svsC3L-00093-26.svs -
Yama Oluşturma: Bu aşamada, histopatoloji görüntülerinden yamalar oluşturulmuştur. Her bir yama, 512x512 piksel boyutunda elde edilmiştir. Ardından yamalardan, embedding vektörleri elde etmek için 224x224 piksel boyutuna yeniden boyutlandırılmıştır.
-
Embedding Elde Etme: Bu aşamada, Google Path Foundation modeli kullanılarak histopatoloji görüntülerinden embedding vektörleri elde edilmiştir. Bu vektörler, görüntülerin özet temsillerini içerir.
Elde edilen verisetine huggingface ile erişmek için bu bağlantıyı kullanabilirsiniz.
Projede kullanılan model yaklaşımları şunlardır:
Autoencoders ile düşük boyutlu bir kodlama alanında görüntü temsili öğrenilmiştir. Kullanılan temel bileşenler:
- Encoder: Konvolüsyonel katmanlar ile veri sıkıştırma.
- Latent Space: Görüntünün özet bilgilerini içeren vektör temsili.
- Decoder: Sıkıştırılmış veriden orijinal görüntüyü yeniden üretme.
GAN'ler, sentetik histopatoloji görüntüleri üretmek için kullanıldı. Kullanılan bileşenler:
- Generator: Rastgele gürültü vektörlerinden gerçekçi görüntüler üretir.
- Discriminator: Gerçek ve üretilmiş görüntüler arasındaki farkı ayırt etmeye çalışır.
- Loss Function: Minimax kayıp fonksiyonu kullanılarak model eğitildi.
Difüzyon modelleri ile daha yüksek kaliteli ve detaylı görüntüler üretmek amaçlanmıştır.
- Gaussian Noise eklenerek eğitim süreci başlatılmıştır.
- Aşamalı olarak gürültü azaltılarak gerçekçi görüntü üretilmiştir.
pip install -r requirements.txtpip install -e .